大量のバイオデータを活用するためのMLOpsの取り組みと課題
バイオとITの融合企業であるCOGNANOで主に機械学習の基盤整備を担当しているtokibiです。この記事では、私のようなソフトウェア開発を中心にキャリアを積んできたITエンジニアが、COGNANOでどのような業務を行っているかのご紹介もかねて、機械学習のワークフロー改善の中で取り組んでいる内容についてお話できればと思います。
COGNANOにおける機械学習の取り組み
COGNANOでは、アルパカが生成するVHH抗体の情報を元に教師データを作成し、抗原抗体の相互作用を予測する深層学習モデルを構築するという試みに取り組んでいます。2010年にVHH抗体の可能性に着目して以来、バイオチームでは長年に渡ってデータを収集してきました。アルパカという生体の免疫反応が反映された情報は、現在データ化されているだけでも3億ほどのレコード数に上り、機械学習に適用するにあたって十分な量が蓄積されています。機械学習に関するより詳しい解説は、研究開発を担当している同僚のつるべーさんが記事を公開しておりますので、ぜひ以下のリンクも併せてご覧ください。
MLOpsの実践に向けた取り組み
機械学習エンジニアとデータサイエンティストが、モデルの性能向上につながる業務を円滑に行えるように、ITチームでも環境の整備を通して支援していく必要があります。
MLOpsの導入について
機械学習におけるワークフロー全体の効率を向上させるための実践手法として、近年ではMLOpsと呼ばれる概念が現れていますが、COGNANOでもこのような原則に則りながら各プロセスの改善を試みているところです。
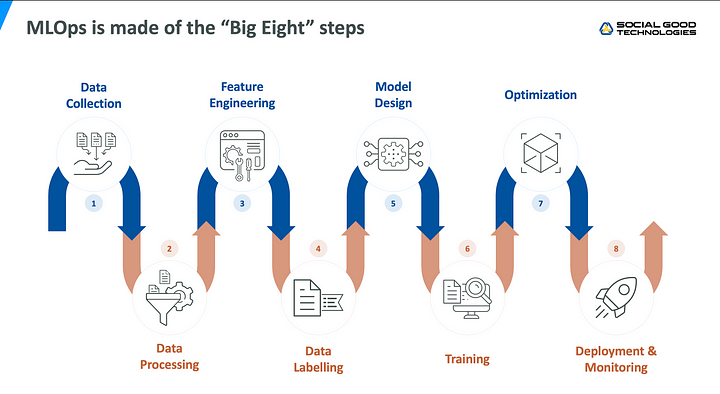
MLOpsは教師データのベースとなるデータの収集や前処理から、モデルのデプロイや運用開始後のモニタリングなども含めて対象とする総合的な概念ですが、今回は、その中の一部であるデータ前処理のプロセスに焦点を当てて、実際に行っている取り組みをお話したいと思います。
VHH抗体を対象にしたデータ前処理の前提
先程は「VHH抗体の情報を元に教師データを作成する」と一言で言ってしまいましたが、機械学習で利用可能な状態にするためには、いくつかの工程を経なければなりません。これには大きく分けると2つの工程が存在し、前半は次世代シーケンサーの出力に対する加工処理で、後半は抗原とVHH抗体のアミノ酸配列のペアに対して正解ラベルを付与していく処理です。
アルパカから採取した抗体遺伝子は、バイオチームによって行われる実験を経て次世代シーケンサーにかけられ、最終的にはFASTQフォーマットで記述されたファイルとして出力されます。この中には、読み取ったDNAの塩基配列と、各配列文字の品質スコア(確からしさ)が記載されています。
出力された直後のFASTQファイルには、不要な配列や品質の低い配列も含まれるため、はじめにアダプタートリミングやクオリティトリミングといった処理を行ったり、後続の処理のために塩基配列からアミノ酸配列に変換するなどの処理を行います。これらは cutadapt や Trimmomatic のようなバイオインフォマティクスの分野で活用されている複数のコマンドラインツールを組み合わせて実現しており、基本的には入力と出力を各コマンドで繋いでいくパイプライン的なプロセスです。

こうしたアミノ酸配列は、バイオチームによって行われる変異体(ウイルスの新型変異タンパク質など)を用いたバイオパニングの度に生成されます。COGNANOでは、バイオパニングによって作成された複数のアミノ酸配列群を独自のアルゴリズムで比較することで、抗原と抗体の結合の有無を検証してラベリングを行っています。ラベリングの詳細については本記事の主題から外れるため省略しておりますが、将来的に投稿される論文の中で解説される予定です。
現状の課題と今後の取り組み
ここまでにお話したデータ前処理のプロセスは、部分的に見ればある程度の自動化は行われていますが、ラベリングの一部では表計算ツールを用いて手動で行っている箇所もあります。また、実験によって新しい抗体情報が追加される度に、データの前処理を行い、過去の情報も含めて教師データを作成した上で、学習にかけて作成されたモデルの性能を評価するという一連の流れに関しても、それぞれの担当者が手動で行っているのが現状です。その中で浮き彫りになってきた課題と今後の取り組みは以下の2つです。
1. ドメイン知識とIT技術の融合
先に述べたワークフロー全体の課題の他に、個別のプロセスの中で使用されているパラメータや手法についても改善の余地があります。現在はバイオチームのドメイン知識を元に選定されていますが、より良い選択肢が存在する可能性は十分に考えられますので、将来的には網羅的な比較検討も必要になるでしょう。このような課題の改善に向けて、現在はMLOpsの前身となるDevOpsの原則に基づいて、次のような基本的な環境の構築から始めているところです。
- データの保管場所にクラウドのストレージを利用することで、アップロードなどのイベントをトリガーに必要な処理を呼び出せるようにする
- 実行環境をコンテナ化することで、再現性や可搬性を向上させる
- ワークフロー自動化のためのパイプラインを構築する
また、今後はモデル開発環境の整備や、パラメータおよび手法がモデル性能に与える影響の評価といった機械学習特有のプロセスに対応していく必要もあります。そのために並行して、Kubeflowのようなオープンソースのプロジェクトや、メガクラウドの各社が提供するMLOpsの実行を総合的にサポートするプロダクト等の導入も視野に入れています。
このようなプロダクトの導入にあたっては、処理をPythonなどのコードに移行することで、修正のイテレーションを回しやすくなる場面もあると考えています。現状、複数のコマンドラインツールに依存している部分に関しては、実行環境のコンテナ化という選択肢を取っていますが、必要に応じて実行方法の移行も進めていく予定です。
2. エンジニアがバイオに関するドメイン知識を習得すること
加えて、バイオ領域の知識の獲得も必要になると考えています。私自身はこれまでソフトウェア開発を中心に行ってきたエンジニアですので、バイオやバイオインフォマティクスの分野について専門的な知識を有しているわけではありません。そのため、各プロセスの改善に取り組む中で、処理の意図がわからない部分も多数出てきます。幸いにも、Web会議やSlackで同期、非同期を問わず専門家へ気軽に質問できる環境ですので、困ることはありませんが、細部の品質を上げて、より良い基盤環境を構築するためには、私自身も一定のドメイン知識を身につける必要があるでしょう。
COGNANOのバリューには「越境と融合」があり、互いの専門性を尊重しつつも、可能な限り歩み寄るという姿勢を重要視しています。これはバイオとITの領域を両者が越えようとすることに他ならず、そこから生まれる成果が大きな価値を生み出すと考えているためです。この記事で挙げている深層学習モデルの構築も、その中から生まれたものの一つです。現在進行中のプロジェクトを通じて、社会に対してより良い貢献ができるように、日々取り組んでいきたいと思っています。
まとめ
今回はCOGNANOにおける機械学習の前処理工程と、それをMLOpsの枠組みに当てはめていくための取り組みをご紹介しました。最後に社会貢献のような固い話を挟んでしまいましたが、私自身は日々楽しみながら未知の分野に関する学習を重ね、改善に取り組んでいます。
我々が現在利用しているデータセットや、前処理の詳細については、論文の投稿等に併せて公開する予定です。できる限り再現性を高めた状態で提供できるようにしたいと考えておりますので、興味をお持ちいただいている方は、楽しみにお待ちいただければと思います。
最後までお読みくださり、ありがとうございます。COGNANOに関するお話が何かありましたら、社長の伊村さんやCTOのまつもとりーさん、tokibiまでご連絡ください。